本節課程,需要完成擴增子分析解讀1質控 實驗設計 雙端序列合併和2提取barcode 質控及樣品拆分 切除擴增引物 先看一下擴增子分析的整體流程,從下向上逐層分析 分析前準備 # 進入工作目錄 cd example_PE250 上一節回顧:我們提取barcode,質控及樣品拆分,切除擴增引物,經歷了 ...
本節課程,需要完成擴增子分析解讀1質控 實驗設計 雙端序列合併和2提取barcode 質控及樣品拆分 切除擴增引物
先看一下擴增子分析的整體流程,從下向上逐層分析
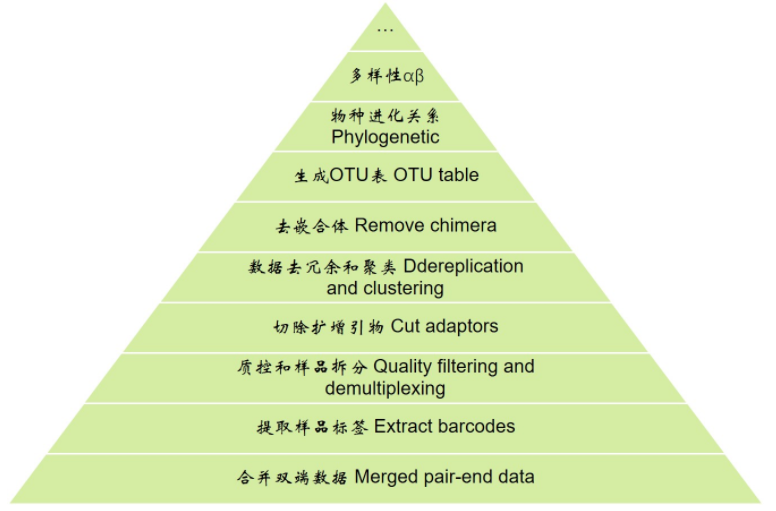
# 進入工作目錄 cd example_PE250上一節回顧:我們提取barcode,質控及樣品拆分,切除擴增引物,經歷了兩節課6步數據處理才拿到我們擴增的高質量目的片段(貌似基因組/RNA-Seq測序結果直接就是這個階段了,可以直接mapping) 接下來我們將這些序列去冗餘、聚類為OTU、再去除嵌合體,這樣就可以獲得高質量的OTU(類似於參考基因組/轉錄組),用於定量分析每個OTU的豐度。這一階段我們使用著名的擴增子分析流程Usearch。 Usearch簡介 http://drive5.com/usearch/ Usearch之前介紹過http://www.cnblogs.com/freescience/p/7270861.html 軟體作者不僅有Usearch一款軟體,它的Muscle(多序列比對,引用18659+4212次),Uparse(OTU聚類演算法,引用1529次), Uchime(擴增子嵌合體檢測,引用3558次)等眾多流行工具,個人引用超4萬次,而且發的軟體大多由作者一人完成,佩服。 Usearch安裝 這個軟體64位版收費,但32位對任何人免費,可以在下麵網址下載 http://www.drive5.com/usearch/download.html 同意許可協議,選擇軟體版本(5.2 — 10.0),選擇運行平臺(Linux, Windows或Mac OSX)填寫郵箱獲得下載地址。不允許私人傳播。 這裡我選擇10.0版本,系統選擇Linux。 收到的郵件中第一個鏈接即下載地址,後面兩個鏈接為幫助文檔和安裝說明,先不用管,按我下麵的操作來。
# 下載程式並重命名:下載鏈接來自郵件,請用戶自行複製郵件中地址替換下麵代碼中的網址;或者在windows裡面下載並重命名為usearch10 wget -O "usearch10" http://drive5.com/cgi-bin/upload3.py?license=XXXXXX # 添加可執行許可權 chmod +x usearch10 # 運行程式測試,成功可顯示程式版本、系統信息和用戶授權信息 ./usearch107. 格式轉換 做生信為什麼要學Python/Perl/Shell這些語言,主要原因是各軟體間要求的具體格式不同,需要進行格式轉換,才能繼續運行。因此想成為高手,不會語言基本寸步難行。 我們現在將QIIME拆分的結果類型,要轉換成Usearch要求的格式。常見的解決思路是讀Usearch幫助看它的格式要求,寫個Python/Perl腳本轉換格式。我這裡使用了Shell腳本一行解決,優點是快,但缺點很多(人不容易看懂、不同Linux系統shell版本不同可能失效) 我們要轉換的序列文件其實一直是fasta格式,只是序列名稱行格式不同
# 目前格式 >KO1_0 HISEQ:419:H55JGBCXY:1:1101:1931:2086 1:N:0:CACGAT orig_bc=TAGCTT new_bc=TAGCTT bc_diffs=0 # Usearch要求的格式 >KO1_0;barcodelabel=KO1;
# 格式轉換 sed 's/ .*/;/g;s/>.*/&&/g;s/;>/;barcodelabel=/g;s/_[0-9]*;$/;/g' temp/PE250_P5.fa > temp/seqs_usearch.fa上面這條命令有點複雜。sed是linux的一條命令,又是一種語言,擅長文本替換。替換的思路分四步:首先s/ ./;/g將原文件空格後面的內容(全是無用信息)替換為分號;其次s/>./&&/g是將序列名重覆一次;再次s/;>/;barcodelabel=/g將重覆後的;>替換為;barcodelabel=;最後s/_[0-9]*;$/;/g替換序列編號為分號。這隻是我的思路,分析數據如解答數學題,可以有多種解法,你夠聰明還會想出更好的解法。 新人一定感覺這命令每句都不像人話,我告訴你Perl和Shell就是這樣—難讀但高效。改用易讀的Python語言,肯定沒有Shell簡潔。 8. 去冗餘 為什麼要去冗餘? 因為原始序列幾百萬條,聚類計算的時間極其恐怖。而已知擴增子測序結果中序列重覆度高,並且大量出現1次或幾次的序列統計學和功能上意義不大。因此將幾百萬條序列去冗餘,並過濾低豐度序列,一般只剩幾萬條,極大的減少了下游分析的工作量,並可使結果更容易理解。 usearch10的去冗餘命令叫-fastx_uniques,緊跟著輸入文件; -fastaout 接輸出文件; -minuniquesize 參數設置保留的最小豐度reads數,建議最小設置為2,去掉所有的單次出現序列(singletons),數據量大建議設置總數據量的百萬分之一併取整數部分 -sizeout 在序列名稱中添加序列出現的頻率
# 序列去冗餘 ./usearch10 -fastx_uniques temp/seqs_usearch.fa -fastaout temp/seqs_unique.fa -minuniquesize 2 -sizeout計算過程中出現如下信息:
00:06 607Mb 100.0% Reading temp/seqs_usearch.fa 00:06 574Mb CPU has 96 cores, defaulting to 10 threads 00:08 915Mb 100.0% DF 00:09 935Mb 1268345 seqs, 686530 uniques, 624363 singletons (90.9%) 00:09 935Mb Min size 1, median 1, max 18774, avg 1.85 62167 uniques written, 182874 clusters size < 2 discarded (26.6%)主要內容為讀取輸入文件; 檢查到系統有96個CPU,預設使用了10個線程; 總共有1268345條序列,其中非重覆的序列有686530個,非重覆且只出現一次的有624363個(90.9%的非冗餘序列是singletons,多嗎?); 最小值、中位數、最大值、平均值;輸出結果有62167個結果,丟棄掉的數據占26.6%。 本條命令的詳細使用,請閱讀官方文檔 http://www.drive5.com/usearch/manual/cmd_fastx_uniques.html 9. 聚類OTU 為什麼要聚類OTU? 是因為Unique的序列仍然遠多於物種數量,並且擴增的物種可能存在rDNA的多拷貝且存在變異而得到來自同一物種的多條序列擴增結果。目前人為定義序列相似度通常97%以上為OTU,大約是物種分類學種的水平,實際上1個OTU可能包括多個物種,而一個物種也可能擴增出多個OTU。 下麵我們用usearch10將非冗餘的序列聚類 -cluster_otus接輸入文件; -otus後面為輸出的otu文件的fasta格式; -uparseout輸出聚類的具體細節 -relabel Otu為重命名序列以Otu起始
# 聚類OTU ./usearch10 -cluster_otus temp/seqs_unique.fa -otus temp/otus.fa -uparseout temp/uparse.txt -relabel Otu程式運行過程會顯示運行時間、進度,發現的OTU,以及嵌合體數據;結果如下:
04:11 84Mb 100.0% 5489 OTUs, 9209 chimeras程式一共運行了3分39秒,聚類發現5486個OTUs,同時發現了9187個嵌合體並已被丟棄。 Usearch聚類演算法之所以能發表在Nature Method上,就是因為其演算法UParse在非常強的嵌合體檢測能力,對人工重組數據評估,更接近真實結果。下一節我們將詳細講嵌合體產生的原因,以及去除的原理。 本條命令的詳細使用,請閱讀官方文檔 http://www.drive5.com/usearch/manual/cmd_cluster_otus.html 小技巧:統計fasta文件中序列的數量 fasta文件每條序列以大於號(>)開始,其數量與序列數量相同,使用grep檢索含有>的行,同時用-c參數對數量進行統計,即可快速獲得fasta文件中序列數量。
# 查看OTU數量 grep '>' -c temp/otus.fa



