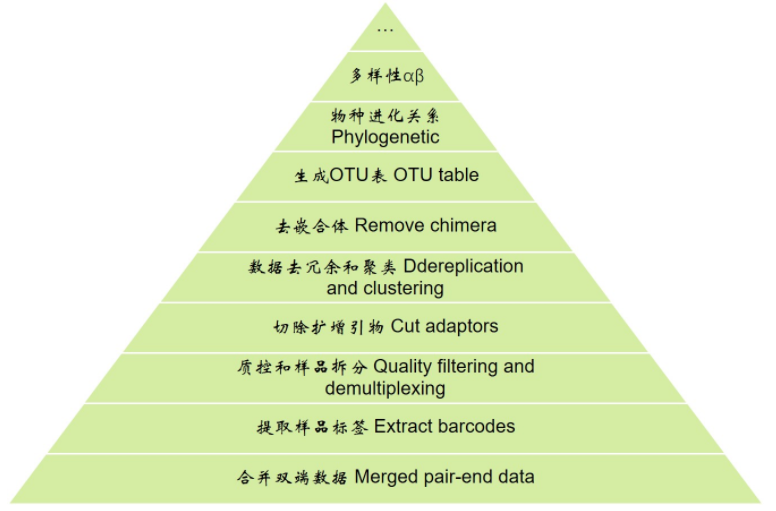
本節課程,需要先完成 擴增子分析解讀1質控 實驗設計 雙端序列合併 2提取barcode 質控及樣品拆分 切除擴增引物 3格式轉換 去冗餘 聚類 先看一下擴增子分析的整體流程,從下向上逐層分析 分析前準備 # 進入工作目錄 cd example_PE250 上一節回顧:我們製作了Usearch要求格 ...
本節課程,需要先完成 擴增子分析解讀1質控 實驗設計 雙端序列合併 2提取barcode 質控及樣品拆分 切除擴增引物 3格式轉換 去冗餘 聚類 先看一下擴增子分析的整體流程,從下向上逐層分析
 分析前準備
分析前準備
# 進入工作目錄 cd example_PE250上一節回顧:我們製作了Usearch要求格式的Fasta文件,對所有序列進行去冗餘和低豐度過濾,並聚類生成了OTU。 接下來我們對OTU進一步去除嵌合體,並生成代表性序列和OTU表。 什麼是chimeras(嵌合體)? 嵌合體序列由來自兩條或者多條模板鏈的序列組成,示意圖如下:
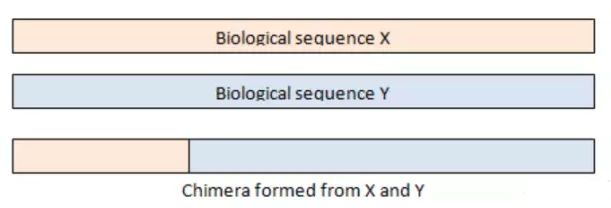 在PCR反應中,延伸階段由於不完全延伸,就會導致嵌合體序列的出現,以上圖為例,在擴增序列X的過程中,在序列延伸階段,只產生了部分X序列延伸階段就結束了,在下一輪的PCR反應中,這部分序列作為序列Y的引物接著延伸,擴增就會形成X和Y的嵌合體序列;
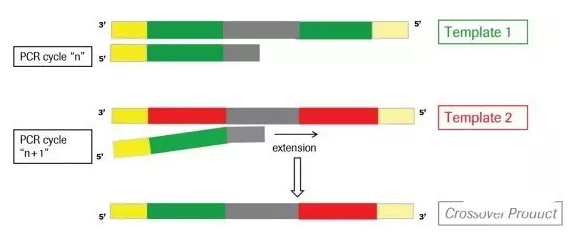
在放一張具體一點的示意圖,不完全延伸產生的序列作為下一輪PCR反應的產物,進行延伸
在PCR反應中,延伸階段由於不完全延伸,就會導致嵌合體序列的出現,以上圖為例,在擴增序列X的過程中,在序列延伸階段,只產生了部分X序列延伸階段就結束了,在下一輪的PCR反應中,這部分序列作為序列Y的引物接著延伸,擴增就會形成X和Y的嵌合體序列;
在放一張具體一點的示意圖,不完全延伸產生的序列作為下一輪PCR反應的產物,進行延伸
 通常在PCR過程中,大概有1%的幾率會出現嵌合體序列,在16S/18S/ITS 擴增子測序的分析中,系統相似度極高,嵌合體可達1%-20%,需要去除嵌合體序列。
嵌合體的比例與PCR迴圈數相關,迴圈數越高,嵌合體比例越高。
有玩過魔獸有小伙伴記得精靈族的終極兵種雙頭龍奇美拉嗎?它的英文就是chimera,即中文的嵌合體,奇美拉是音譯。
10. 基於資料庫去嵌合體(可選)
上文第9步,聚OTU時,已經按照組內的序列相似情況,直接denovo去除了大量嵌合體。目前這步基於資料庫去嵌合體,在以前的分析中是必做的,但隨著技術發展,發現這步可能也會造成假陰性。讀者可以實驗設計、初步結果和預期來判斷是否需要這步處理。本文示例對每一步均進行操作,即是個人風格,又是為了給大家展現一個比較全面的流程。之前Usearch作者推薦使用RDP數據去嵌合,並提供了下載鏈接;現在作者建議,如果做,就用Sliva或Unite這種全面的大資料庫,不推薦用RDP這種小資料庫,以前的建議是錯的。軟體方法均是不斷進步的,我還沒有系統比較作者的新建議有多大改進,這裡還是按照原來的方法進行,讀者可以自行嘗試新方法。
通常在PCR過程中,大概有1%的幾率會出現嵌合體序列,在16S/18S/ITS 擴增子測序的分析中,系統相似度極高,嵌合體可達1%-20%,需要去除嵌合體序列。
嵌合體的比例與PCR迴圈數相關,迴圈數越高,嵌合體比例越高。
有玩過魔獸有小伙伴記得精靈族的終極兵種雙頭龍奇美拉嗎?它的英文就是chimera,即中文的嵌合體,奇美拉是音譯。
10. 基於資料庫去嵌合體(可選)
上文第9步,聚OTU時,已經按照組內的序列相似情況,直接denovo去除了大量嵌合體。目前這步基於資料庫去嵌合體,在以前的分析中是必做的,但隨著技術發展,發現這步可能也會造成假陰性。讀者可以實驗設計、初步結果和預期來判斷是否需要這步處理。本文示例對每一步均進行操作,即是個人風格,又是為了給大家展現一個比較全面的流程。之前Usearch作者推薦使用RDP數據去嵌合,並提供了下載鏈接;現在作者建議,如果做,就用Sliva或Unite這種全面的大資料庫,不推薦用RDP這種小資料庫,以前的建議是錯的。軟體方法均是不斷進步的,我還沒有系統比較作者的新建議有多大改進,這裡還是按照原來的方法進行,讀者可以自行嘗試新方法。
# 下載Usearch推薦的參考資料庫RDP wget http://drive5.com/uchime/rdp_gold.fa # 基於RDP資料庫比對去除已知序列的嵌合體 ./usearch10 -uchime2_ref temp/otus.fa \ -db rdp_gold.fa \ -chimeras temp/otus_chimeras.fa \ -notmatched temp/otus_rdp.fa \ -uchimeout temp/otus_rdp.uchime \ -strand plus -mode sensitive -threads 96採用-uchime2_ref參數去嵌合體,後面接OTU序列(輸入文件); -db 指定參考資料庫,這裡用RDP; -chimeras 輸出檢測為嵌合體的序列; -notmatched 輸出不匹配資料庫的結果,即非嵌合,非相同序列; -uchimeout 輸入嵌合體的檢測詳細信息,如每個嵌合體的來源,與那幾個親本相似等; -strand 指定鏈方向,一般為正; -mode 選擇模式,敏感的代價是嵌合體鑒定的高假陽性率; -threads 設計線程數,程式預設系統小於10個線程為單線程;多於10個線程為10線程,根據實際情況設置,不清楚不用更好。 上面計算結果Chimeras 2669/5489 (48.6%), in db 51 (0.9%), not matched 2769 (50.4%),即5489個OTU有2669檢測為嵌合、51個與資料庫序列一致為非嵌合,另外2769與資料庫不匹配不確定是否為嵌合。對應temp/otus_rdp.uchime文件中第三列的Y/N/? 我們想要是的排除嵌合的部分,即51+2769=2820。思路是將全部OTU中鑒定為嵌合的排除掉。
# 獲得嵌合體的序列ID grep '>' temp/otus_chimeras.fa | sed 's/>//g' > temp/otus_chimeras.id # 剔除嵌合體的序列 filter_fasta.py -f temp/otus.fa -o temp/otus_non_chimera.fa -s temp/otus_chimeras.id -n # 檢查是否為預期的序列數量2820 grep '>' -c temp/otus_non_chimera.fa11. 去除非細菌序列(可選) 此步也是非必須的,容易造成假陰性。分析中有很多個人習慣的因素在裡面,所以不同人的分析結果,也會略有不同。也缺少系統的評估到底那些更好,因為好與不好是有條件的,如何判斷也不容易説清楚,這就是經驗;項目經驗是經過大量的項目反覆研究積累出來的。 個人習慣在大數據面前,結果再多也沒用,得找到有意義的東西,所以原則上是能舍即舍,更容易發現規律。萬一沒有發現,再回去把扔掉的撿回來試試。如果什麼都不仍,規律可能永遠藏在大數據的海洋中。 這步的原理是將OTU與Greengene (http://greengenes.secondgenome.com)的Align資料庫比對,篩選序列相似性大於75%以上的序列作為細菌序列;此步可以排除外源非細菌的污染,非細菌序列在接下來的分析中無法註釋物種分類,也很難分析。
# 下載Greengene最新資料庫,320MB wget -c ftp://greengenes.microbio.me/greengenes_release/gg_13_5/gg_13_8_otus.tar.gz # 解壓數據包後大小3.4G tar xvzf gg_13_8_otus.tar.gz # 將OTU與97%相似聚類的代表性序列多序列比對,大約8min time align_seqs.py -i temp/otus_non_chimera.fa -t gg_13_8_otus/rep_set_aligned/97_otus.fasta -o temp/aligned/ # 無法比對細菌的數量 grep -c '>' temp/aligned/otus_non_chimera_failures.fasta # 1860 # 獲得不像細菌的OTU ID grep '>' temp/aligned/otus_non_chimera_failures.fasta|cut -f 1 -d ' '|sed 's/>//g' > temp/aligned/otus_non_chimera_failures.id # 過濾非細菌序列 filter_fasta.py -f temp/otus_non_chimera.fa -o temp/otus_rdp_align.fa -s temp/aligned/otus_non_chimera_failures.id -n # 看我們現在還有多少OTU:975 grep '>' -c temp/otus_rdp_align.fa經過這一步過濾,從2820非嵌合的OTU,只剩下975個與細菌相似的OTU,這種數量才更接近真相。有些研究經常搞幾千、幾萬的OTU,假陽性結果90%以上,你覺得意義何在,如何指導下游實驗。 對於真菌ITS/18S,一般不建議用Unite資料庫去嵌合,因為ITS/18S在所有真核生物中都有,有待物種註釋後進一步確認。 12. 產生代表性序列和OTU表 代表性序列(representative sequences)即為確定的最終版的OTU,類似於參考基因組/cDNA將為索引的字典。然後將所有數據mapping於OTU上來確定各物種的豐度。 OTU表,是每個OTU在每樣品中的豐度值,本質上每種高通量測序結果,都會有一個類似的表,如RNA-Seq是基因表達與樣品的表
# 重命名OTU,這就是最終版的代表性序列,即Reference(可選,個人習慣)
awk 'BEGIN {n=1}; />/ {print ">OTU_" n; n++} !/>/ {print}' temp/otus_rdp_align.fa > result/rep_seqs.fa
# 生成OTU表
./usearch10 -usearch_global temp/seqs_usearch.fa -db result/rep_seqs.fa -otutabout temp/otu_table.txt -biomout temp/otu_table.biom -strand plus -id 0.97 -threads 10
# 結果信息 01:20 141Mb 100.0% Searching seqs_usearch.fa, 32.3% matched
# 預設10線程,用時1分20秒,有32.3%的序列匹配到OTU上;用30線程反而用時3分04秒,不是線程越多越快,分發任務也是很費時間的
現在我們獲得了OTU表,用less temp/otu_table.txt查看一下吧。同時還有biom可處理的標準json格式文件,用於後續分析



