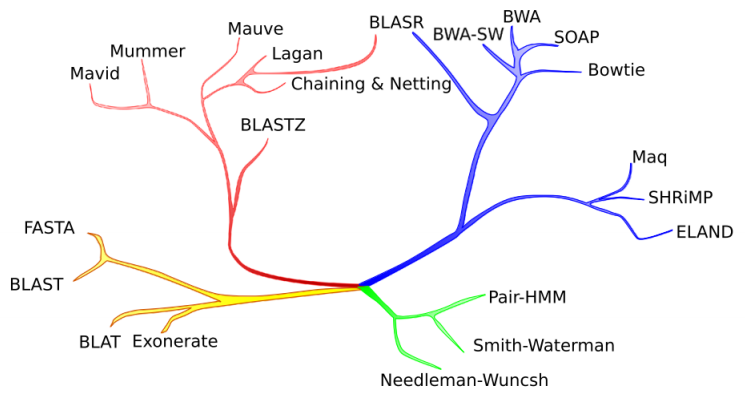
(green) short pairwise alignment / detailed edit model; (yellow) database search / divergent homology detection; (red) whole genome alignment / alignm ...
簡單的局部比對:Blast、Blat等
Blast類型包括以下幾種: Blastn---核酸序列與資料庫中的核酸序列的比對 Blastp---蛋白序列與資料庫中的蛋白序列的比對 Blastx---核酸序列翻譯成蛋白質(six-frame protein)後與資料庫中的蛋白序列進行比對 Tblastn---蛋白質序列與資料庫中的核酸序列進行比對 Tblastx---核酸序列翻譯成蛋白質後與資料庫中的核酸序列翻譯成的蛋白質序列進行比對 BLAST建庫 建庫的過程是建立目標序列的索引文件,所用程式是formatdb,通常我們使用FASTA格式的序列作為輸入。用於建庫的FASTA序列是db.seq,formatdb的基本命令是:formatdb -i db.seq [-options]
常用參數有以下幾個:
-p:選擇建庫的類型,T表示蛋白庫,F表示核酸庫。預設值為T
-o:判斷是否分析序列名並建立序列名索引。T表示建立序列名索引,F表示不建立序列名索引。預設值為F
BLAST命令行:

輸出結果:
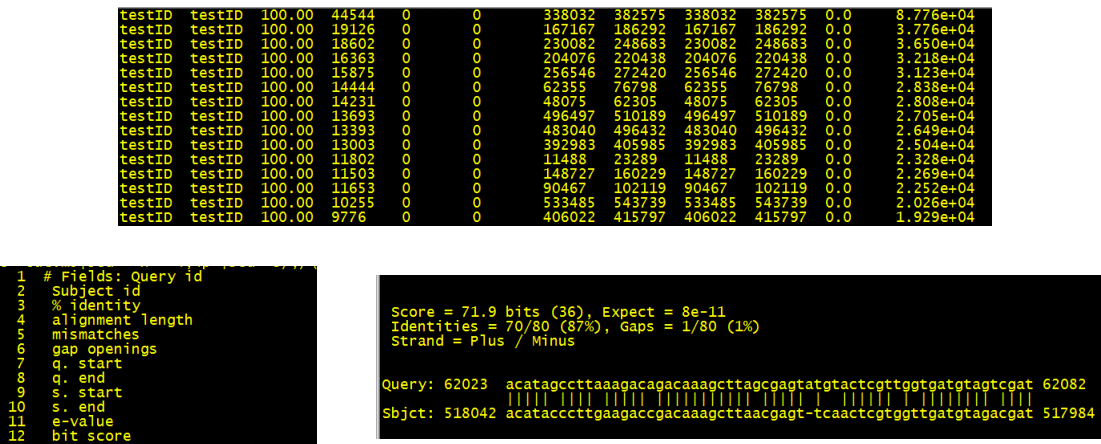
3是相似度、4是能比對上的區域
Blat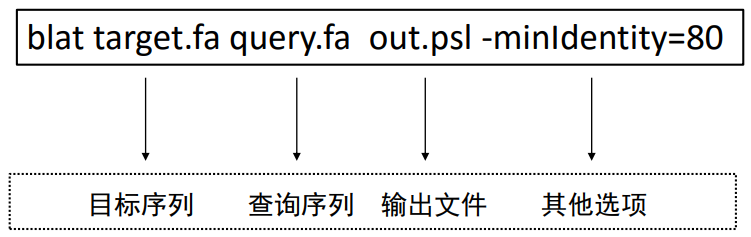
全局比對:MUSCLE、MATT等
muscle -in <inputfile> -out <outputfile>



